TCGA数据库后台是如何规范TCGA样品ID的?
时间:2019-03-01 来源:生信自学网 作者:乐伟
微信公众号:biowolf_cn 点击:次
TCGA数据库现在是生信分析的热点,生信自学网开创了用生信方法解读TCGA数据的先河,给研究者提供了新的研究方案。
有很多学员仍然不是很了解,TCGA所有数据都有的样品ID是如何规范的,那么我们来看看TCGA后台是如何把庞大的数据文本化的?
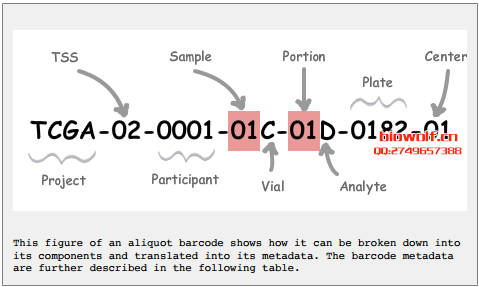
接触和分析过TCGA数据的朋友肯定会经常处理TCGA barcode的前15位(有时12位),实际从上图可以看出TCGA的barcode设计总共有28位之多。
每一个短横杠衔接的都是含不同意义的序列,如下图
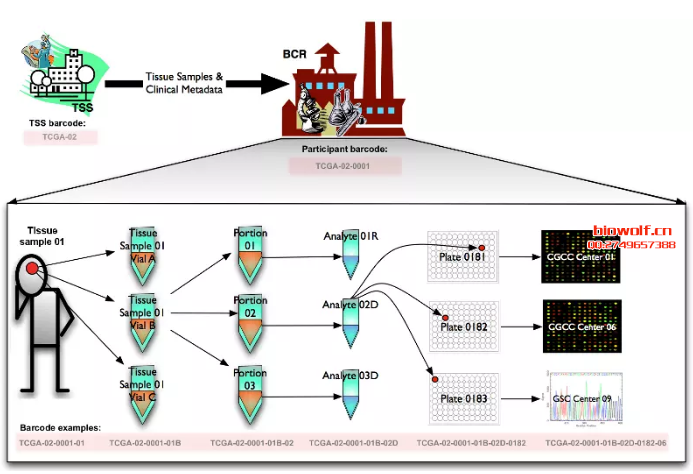 从TCGA数据库我们也找到了相关的说明文档: 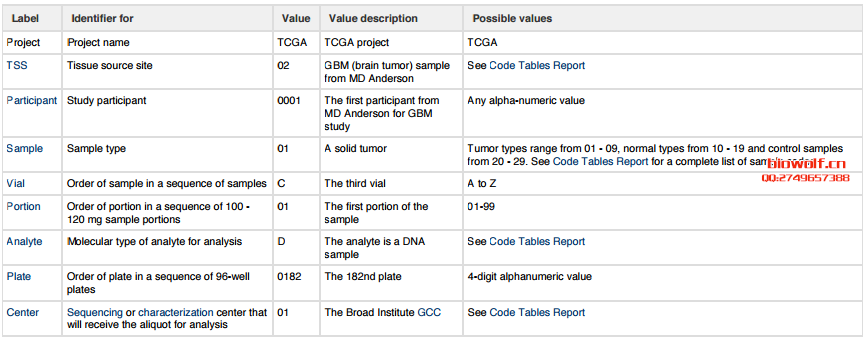
Barcode Types
Barcodes can also be visualized hierarchically, with TSS barcodes at the top of the tree and aliquot barcodes at the bottom. A parent barcode
prefixes any of its descendent barcodes, reflecting the derivation of one biospecimen type from another. For example, samples are collected from
a participant and so the corresponding sample barcodes contain the participant barcode from which they were derived.
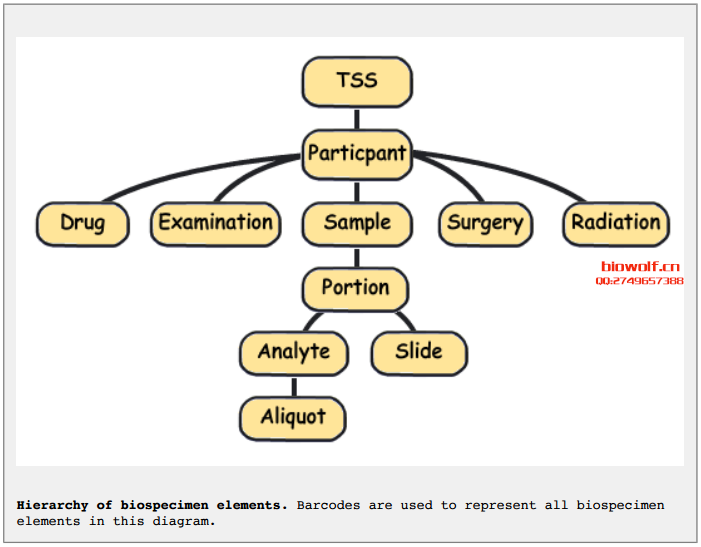
Using the aliquot barcode example from the figure in Reading Barcodes, the following table displays a possible set of related barcodes at each
level of the hierarchy:
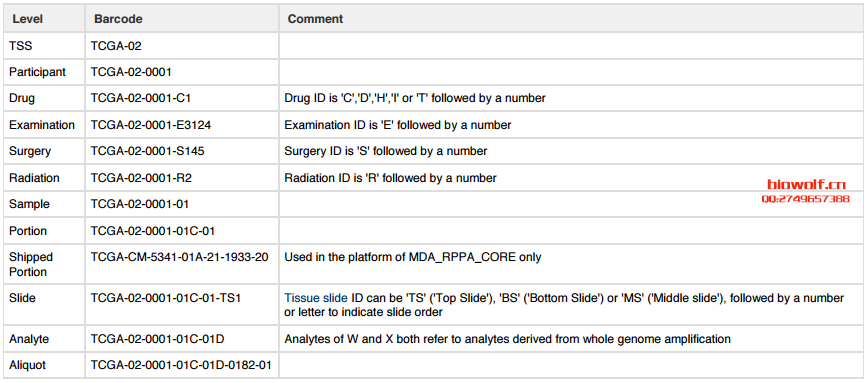
可以看到同一个样本(一个病人的某一个组织块),在实际的实验处理中是分了很多分析试样的,特别是plate部分。这也就导致在实际的分析中有可能会出现多个barcode对应同一个样本(即前15位是一致的)。
通过谷歌引擎找到Biostars上有人对这个问题加以讨论,我按照着提供的链接找到了Broad研究所进行barcode去重的策略:
主要内容如下:
翻译成中文,大致有以下3点:
对于RNA分析, Analyte序列 H>R>T
对于DNA分析,Analyte序列中D>G,W,X
如果经常前面的过滤还重复样本,考虑portion和plate序列,选择更大的
另外,分析不使用福尔马林处理的样本(DNA与RNA分析数据失真,但这一点TCGA已经考虑了)
(责任编辑:伏泽 微信:18520221056) 
|