SNP突变数据整理perl脚本ICGC数据库
时间:2019-07-30 11:26来源:生信自学网 作者:乐伟 点击:
次
从ICGC数据库下载了SNP数据之后,我们需要提取我们需要的数据,整理成突变数据矩阵
得到突变数据之后,数据里面含有病人ID、实验ID等信息,还有我们非常关注的突变影响的基因,原始下载的是ensembl ID,我们希望把ensembl ID转换为我们常见的gene symbol,以便做后续的分析。
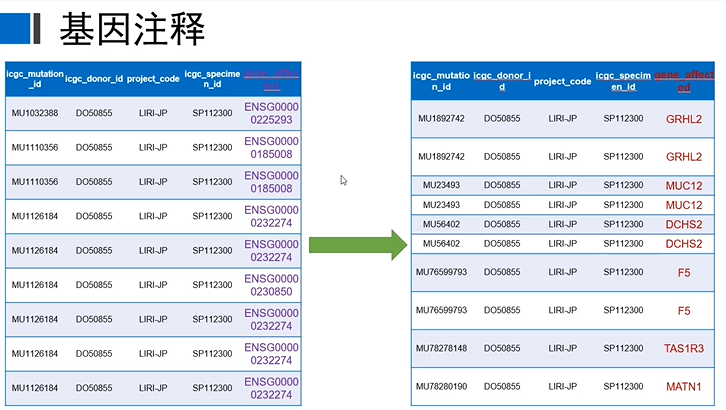
这一步操作,需要实现两个目的,一个是删除没有发生氨基酸改变的突变;一个是ensembl ID转换为gene symbol。操作用到的是perl脚本,脚本是我们生信自学网原创的Perl脚本。
my $gtfFile="human.gtf";
my $outFile="mutation.ann.txt";
my $snpFile=$ARGV[0];
chomp($snpFile);
my %ensemblHash=();
open(RF,"$gtfFile") or die $!;
while(my $line=<RF>)
{
chomp($line);
if($line=~/gene_id \"(.+?)\"\;.+gene_name "(.+?)"\;.+gene_biotype \"(.+?)\"\;/)
{
my $ensembl=$1;
my $symbol=$2;
my $biotype=$3;
$ensemblHash{$ensembl}=$symbol;
}
...
...
获取源码请联系微信:18520221056
或者购买生信自学网课程:
|
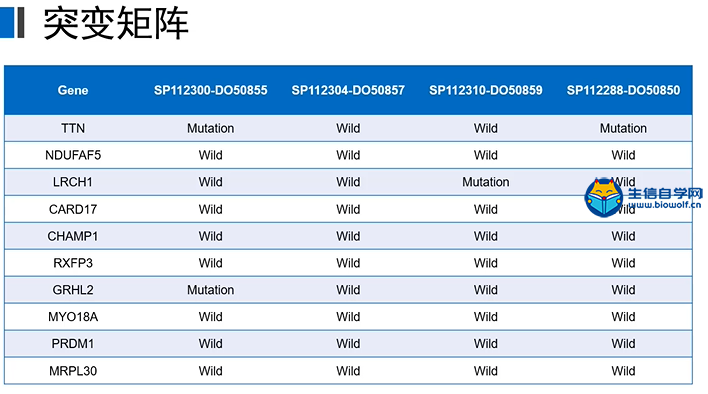
接下需要把突变矩阵提取出来,突变矩阵行名是gene,列名是实验ID和样本ID结合,数据就是突变信息。同时我们也希望知道每个基因在样本突变的频率,也就是在分析的时候,需要做一下统计分析。
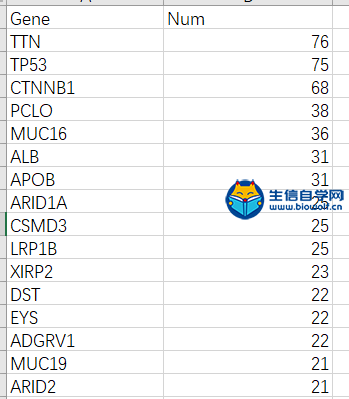
在统计基因突变的表格里,我们看到了几个常见的突变基因,比如TP53,说明生信分析的结果是有研究价值的,也符合已发表的文献和报导。
use strict;
use warnings;
my $file="mutation.ann.txt";
my %hash=();
my @sampleArr=();
my %sampleHash=();
my $gene="all";
my %fieldHash=();
open(RF,"$file") or die $!;
while(my $line=<RF>){
next if($line=~/^\n/);
chomp($line);
my @arr=split(/\t/,$line);
if($.==1){
for(my $i=0;$i<=$#arr;$i++){
$fieldHash{$arr[$i]}=$i;
}
next;
...
...
获取源码请联系微信:18520221056
购买课程获取源码
|
购买ICGC数据库挖掘新课程:
《ICGC数据库挖掘SNP突变数据》

责任编辑:伏泽
作者申明:本文版权属于生信自学网(微信号:18520221056)未经授权,一律禁止转载!
|
BioWolf二维码生成器
------分隔线----------------------------









