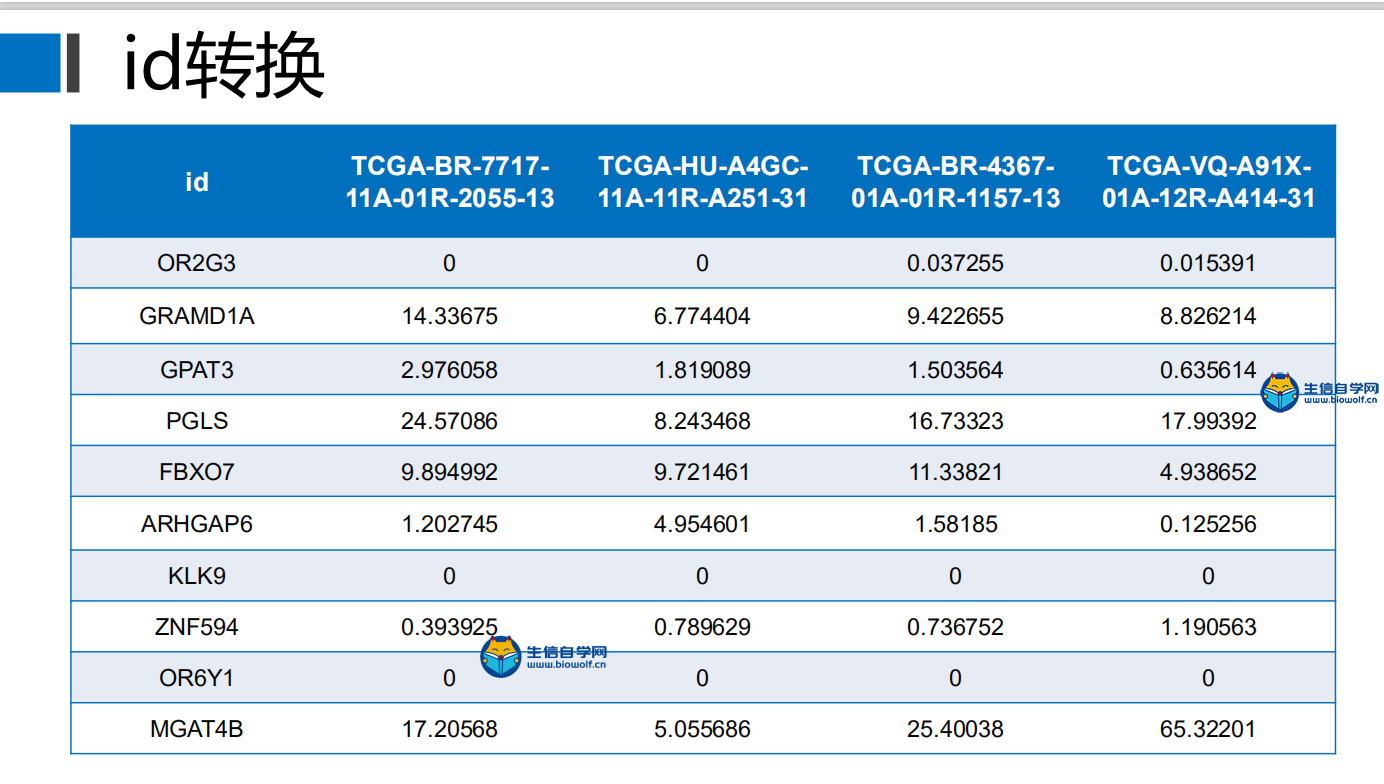
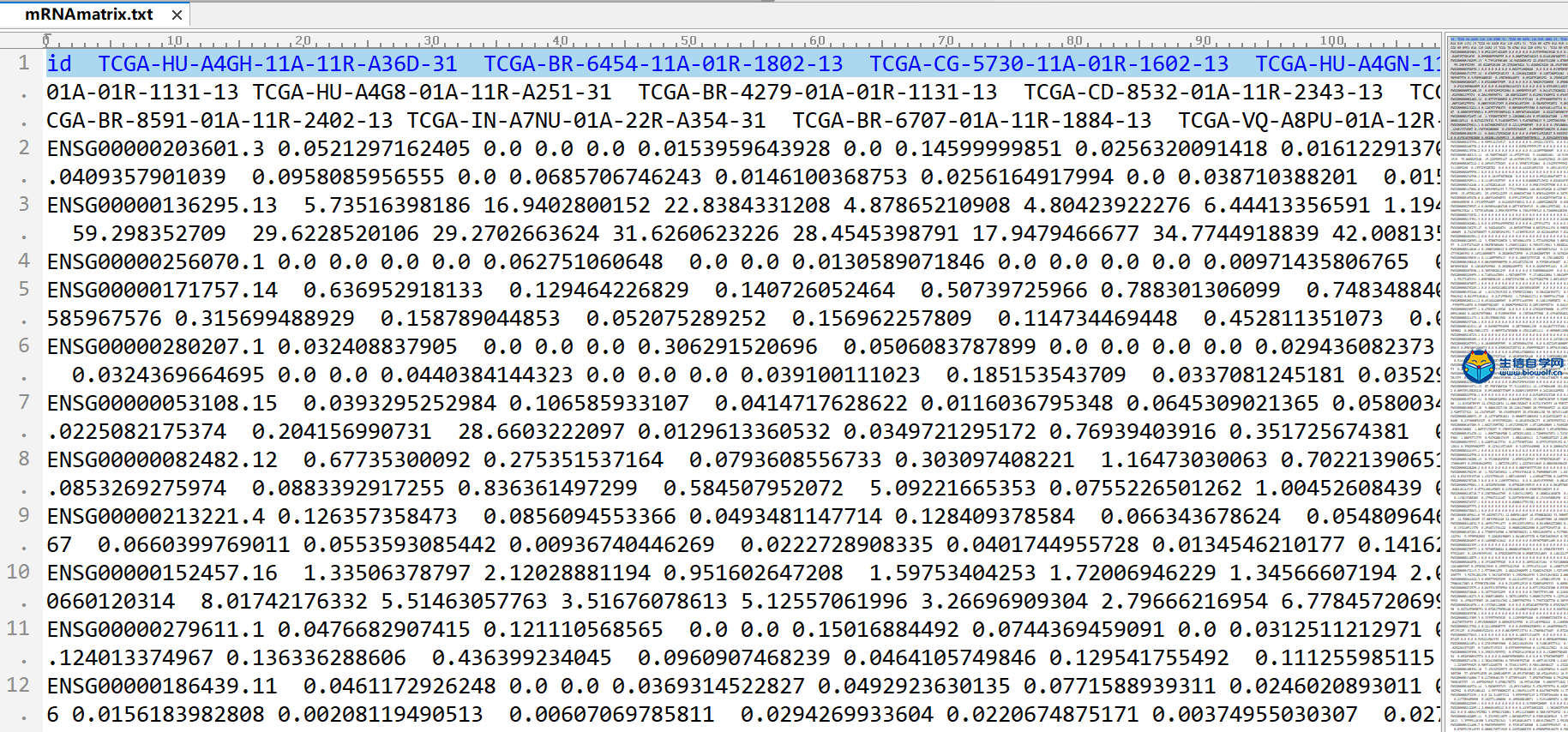
转录组数据的下载及整理TCGA转录组数据下载与整理 我们推荐是使用官网进行下载。我们首先打开浏览器,然后在搜索框里面搜索TCGA gdc,然后进入下载数据的界面。我们进入了界面后,我们要做的第一步就是看我们这个Cart购物车是否为零,如果Cart不为零的话,我们需要把它里面的数据删掉。然后点击Repository进入数据下载界面,接着我们选择我们的疾病类型,比如说我们现在要做胃癌的,我们就选择胃组织,然后选择项目、类型、性别等,这个可以根据自己的需求进行选择下载。 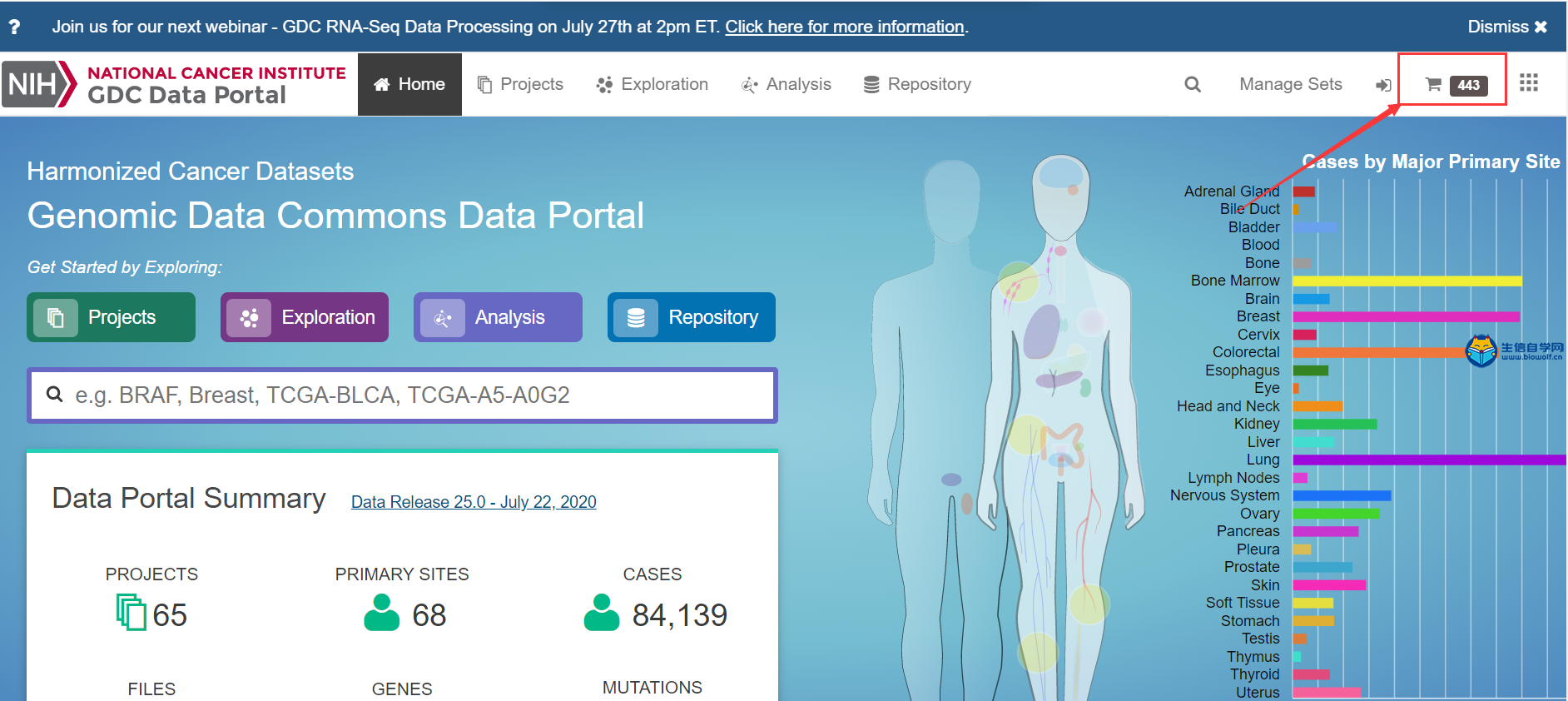 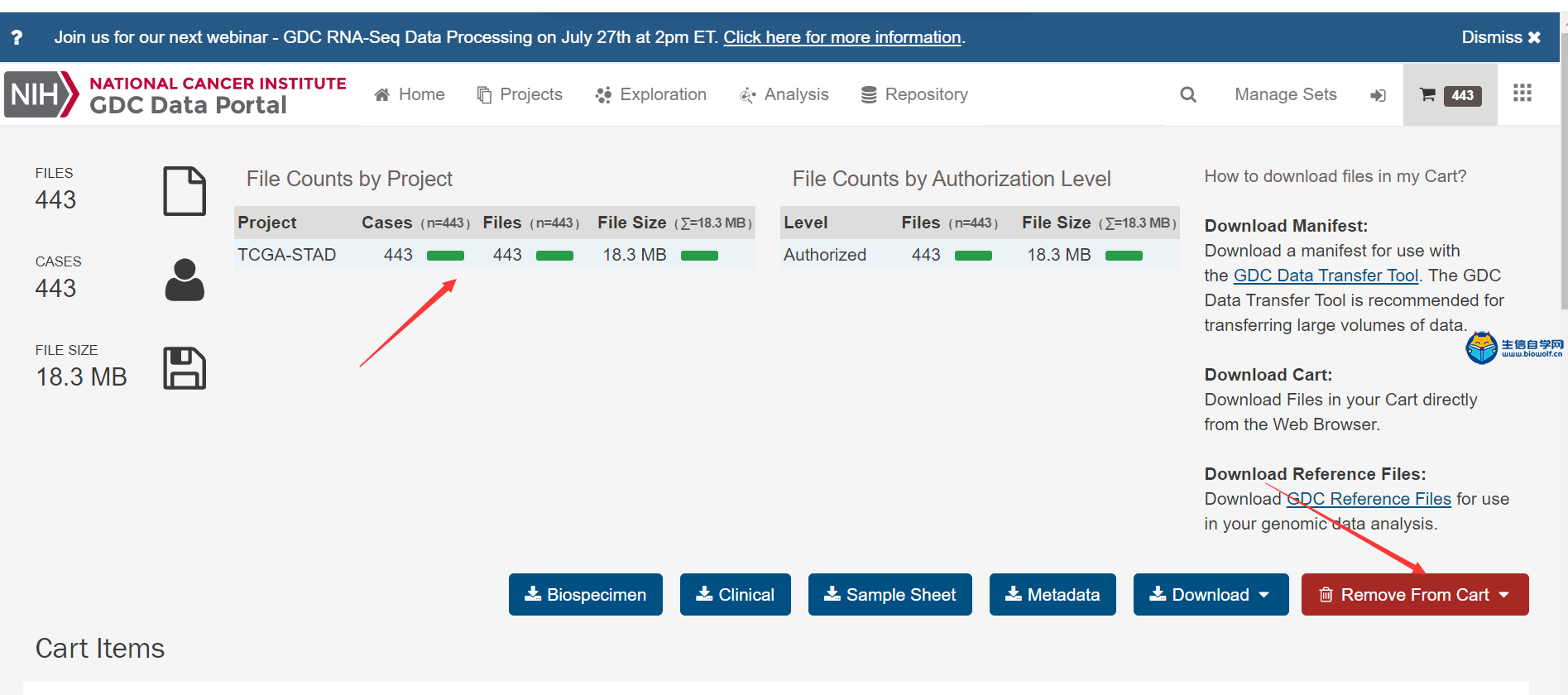  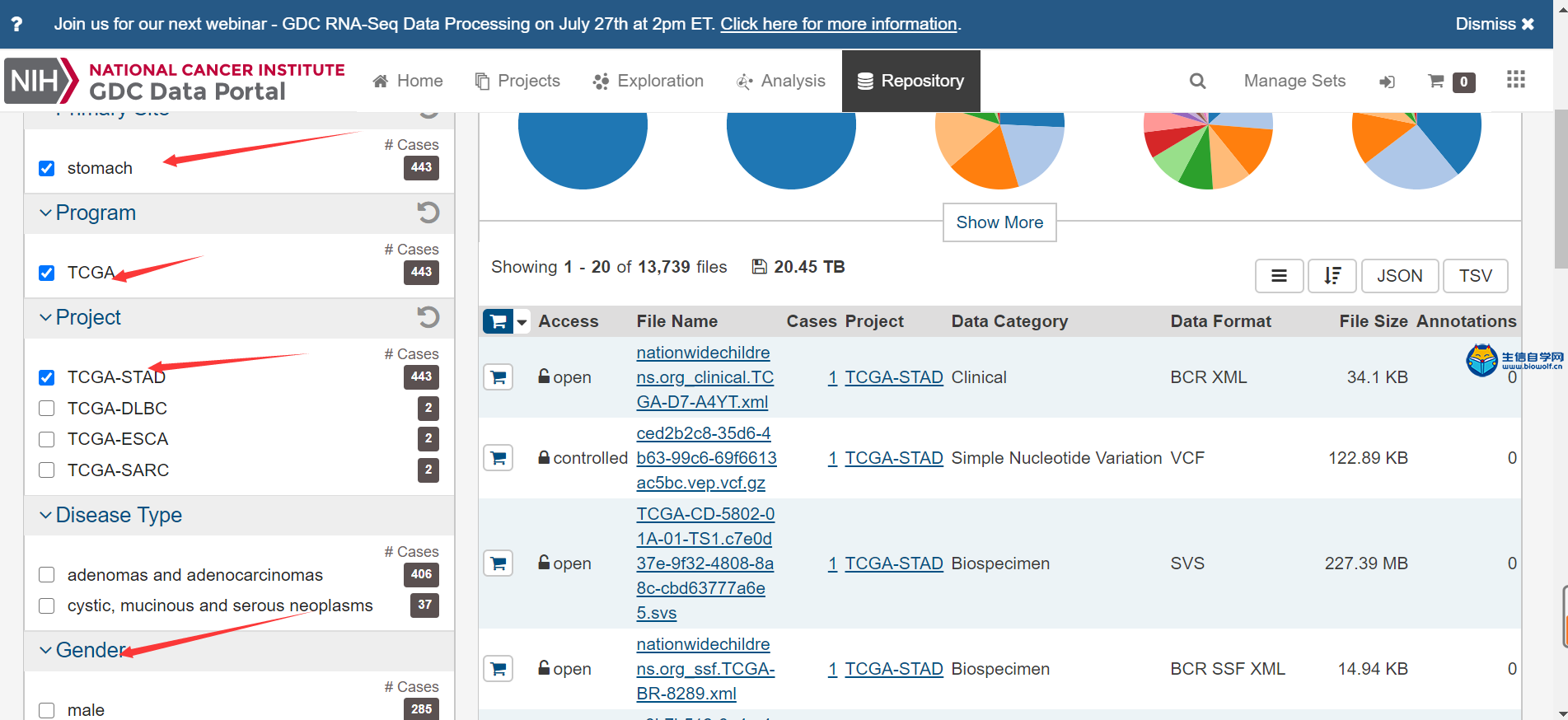 选择好要研究的类型后,接下来我们要选择文件的类型,我们要下载转录组的数据,所以我们选上转录组数据,选择基因的表达量等,然后就可以把选择好后的数据加入到Cart里面下载。我们需要下载几个数据,分别是manifest文件、Cart文件和metadata文件。这样我们的转录组数据就下载好了。 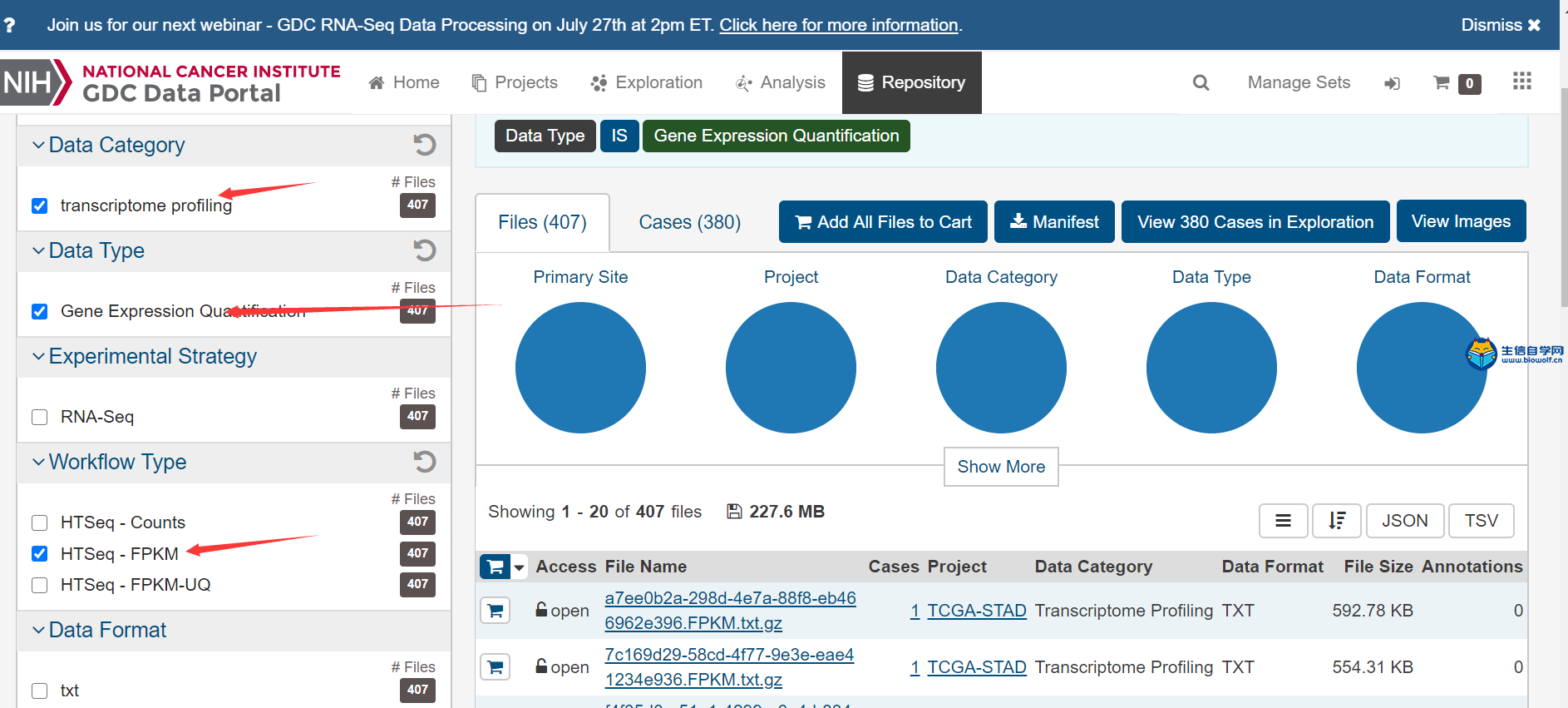 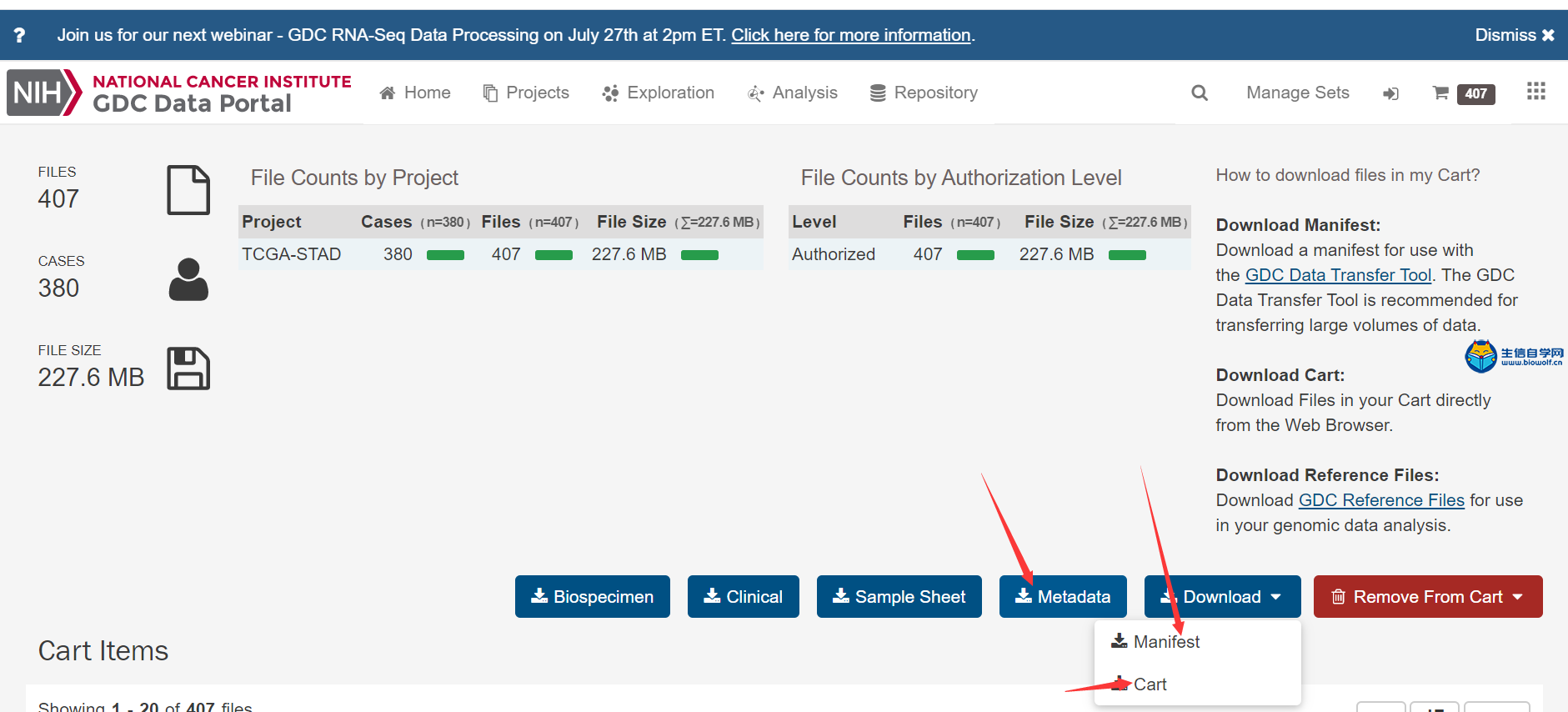 我们下载好了数据后,我们的每个样品都是一个文件,我们需要把它整理成如下的矩阵,这个矩阵的话行名是ID,列名是样品名。同时我们在整理的时候也会把正常样品放前面,把肿瘤样品放后面,方便我们后续的分析。 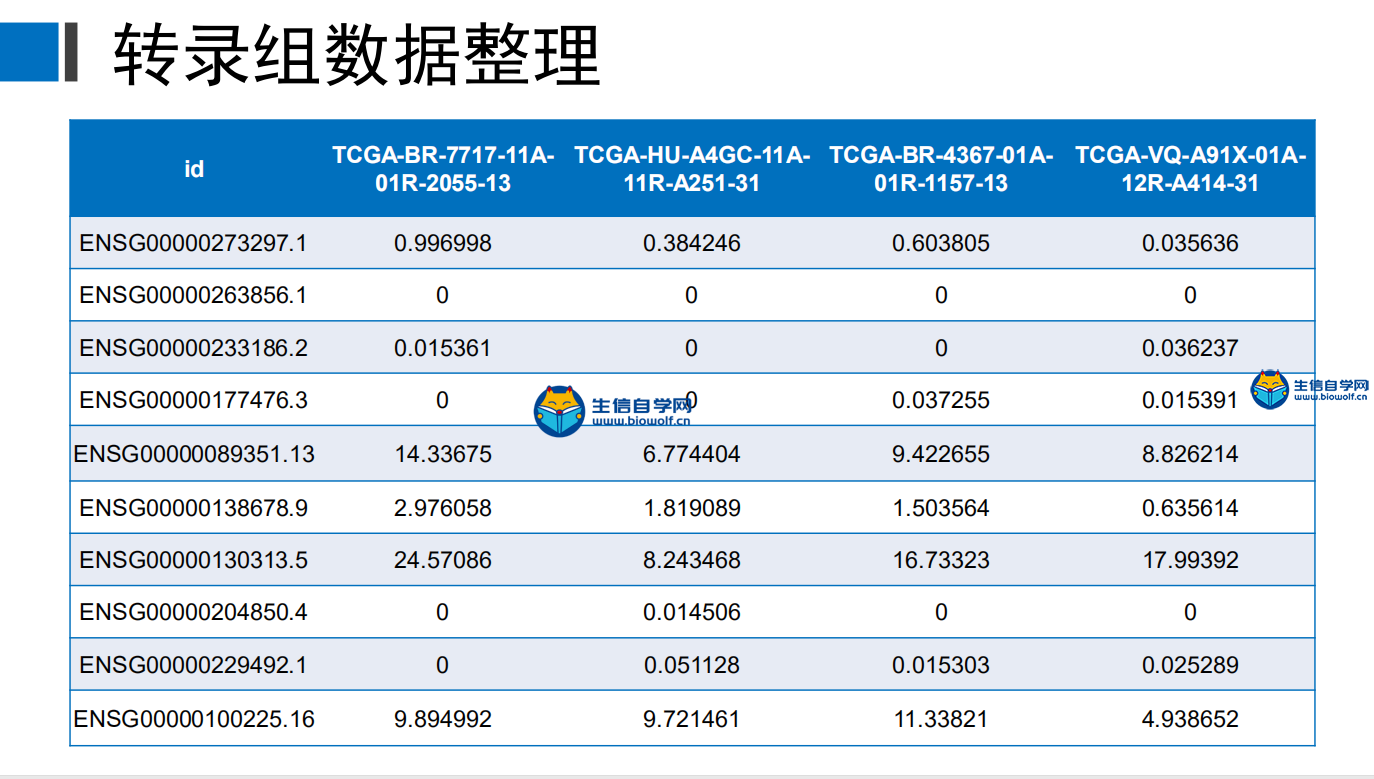 下面我们就对我们下载的数据进行整理,我们需要准备的文件有我们下载好的打包文件和metadata文件,还有我们整理要用的脚本pl文件,我们先将安装包文件解压,然后我们将moveFiles.pl脚本拷贝到解压后的文件夹中,用perl运行该脚本文件,这样我们就可以将每个目录下的文件全部放入同一个目录下。 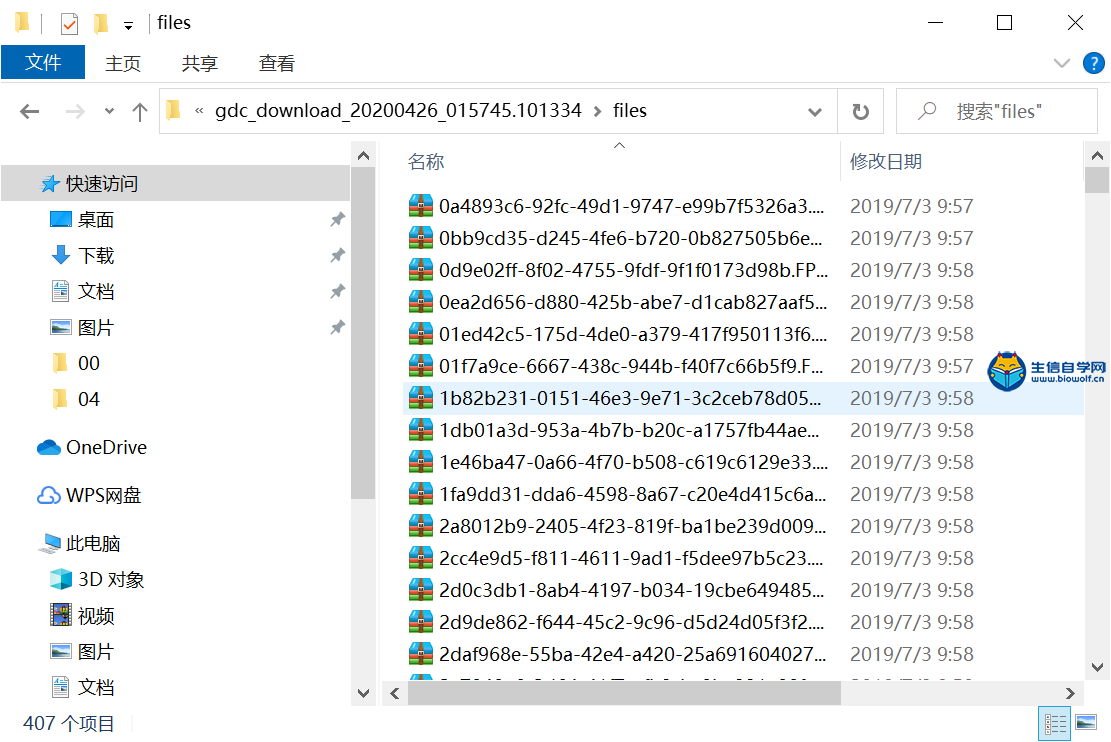 之后我们解压文件里所有的压缩包,等到解压后的文件,接着利用merge.pl脚本将所有的文件进行合并,得到合并后的文件 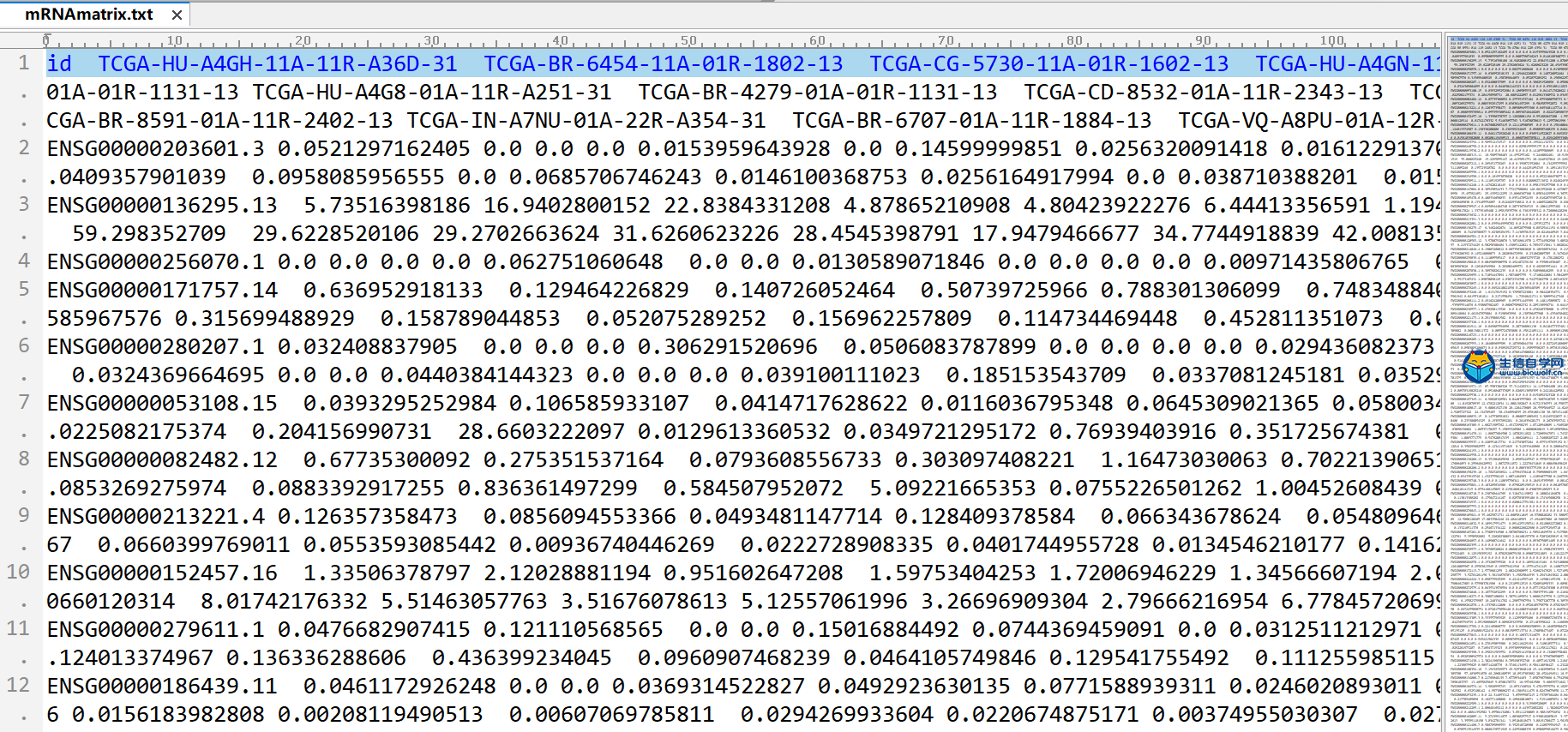
免疫基因对id的转换 精品课程推荐: 《GEO数据库miRNA芯片挖掘》 《甲基化肿瘤分型文章套路视频》 《TCGA肿瘤免疫细胞浸润模式挖掘》 
责任编辑:乐伟 作者申明:本文版权属于生信自学网(微信号:18520221056)未经授权,一律禁止转载! |