|
一、数据库:TCGA 二、内容:下载miRNA表达数据文件,提取miRNA表达矩阵文件 三、癌症数据:宫颈鳞状细胞癌CESC 四、方法: 1、可视化下载txt原始文件 2、perl脚本提取miRNA表达矩阵,为接下来的差异表达做数据准备 五、下载步骤: 1、第一步和“TCGA临床数据下载”的步骤一样,直接参考 2、选择的方法:CASE选项框依次选择——"Primary Site"-Cervix——"Cancer Program"-TCGA——"Project"-TCGA-CESC——其他默认即可 Files选项框依次选择——"Data Category"-Transcriptome Profiling——"Data Type"-miRNA Expression Quantification——其他默认即可 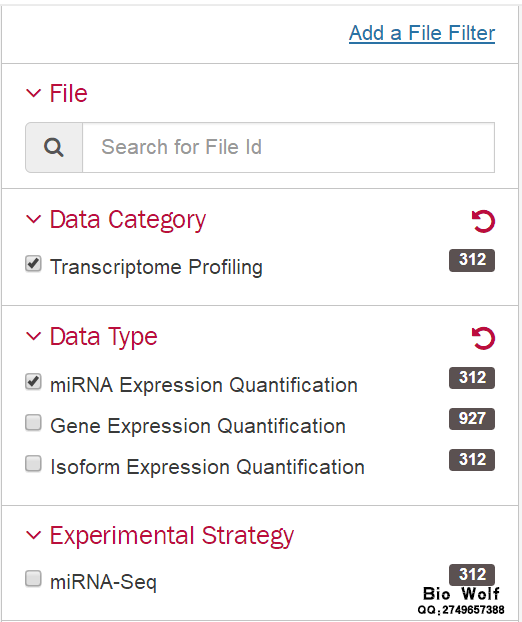 这是右边可以得到Cases数目307个,Files数目312个,大小是15.70M,Cases数目大于Files的数目,说明某些样本有多个表达数据文件。 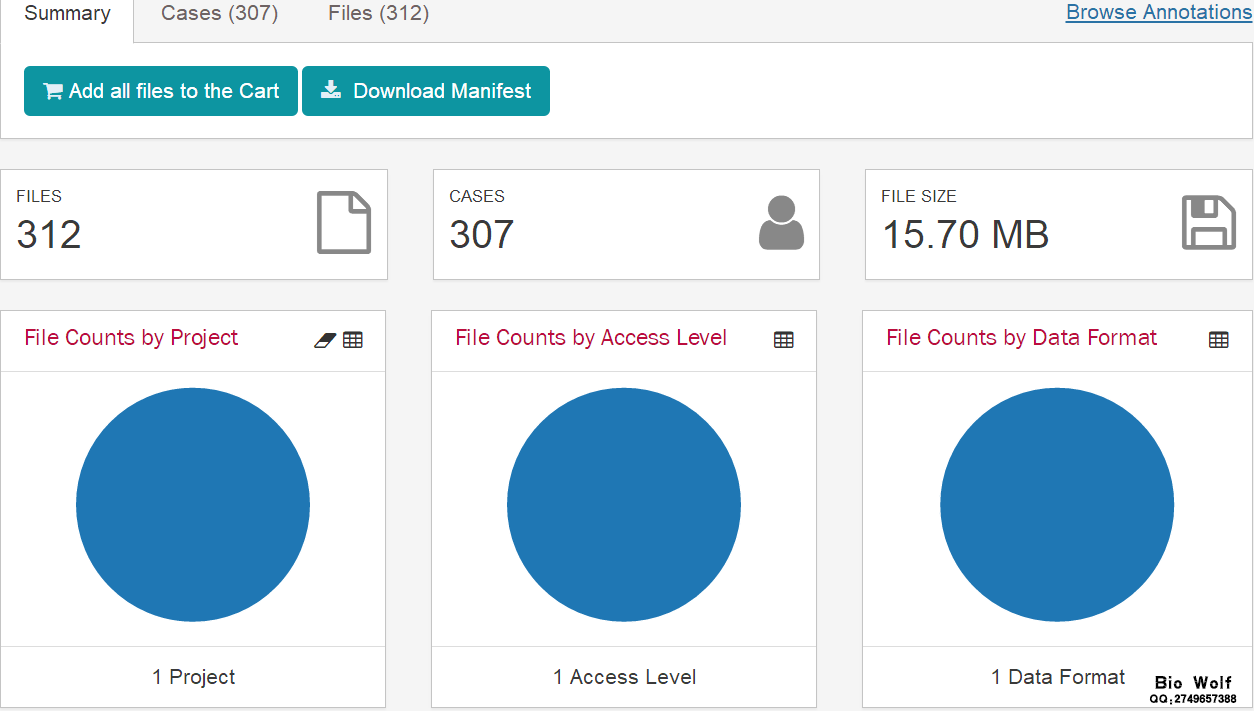 3、点击"Add all files to the cart",然后进入右上角的"Cart"进入数据展示和下载页面 说明:"Cart"是TCGA数据库类似购物车的一个工具,里面是我们选到的数据界面。 4、在“Cart"页面中,我们需要下载3个数据:Metadata、"Download"-Manifest、Cart 说明: Metadata:最后一次随访的临床数据 Manifest:样本注释文件,主要用于Data Transfer Tool工具下载数据时使用 Cart:压缩包,包含所有的txt文件,也就是miRNA表达数据的压缩包文件。 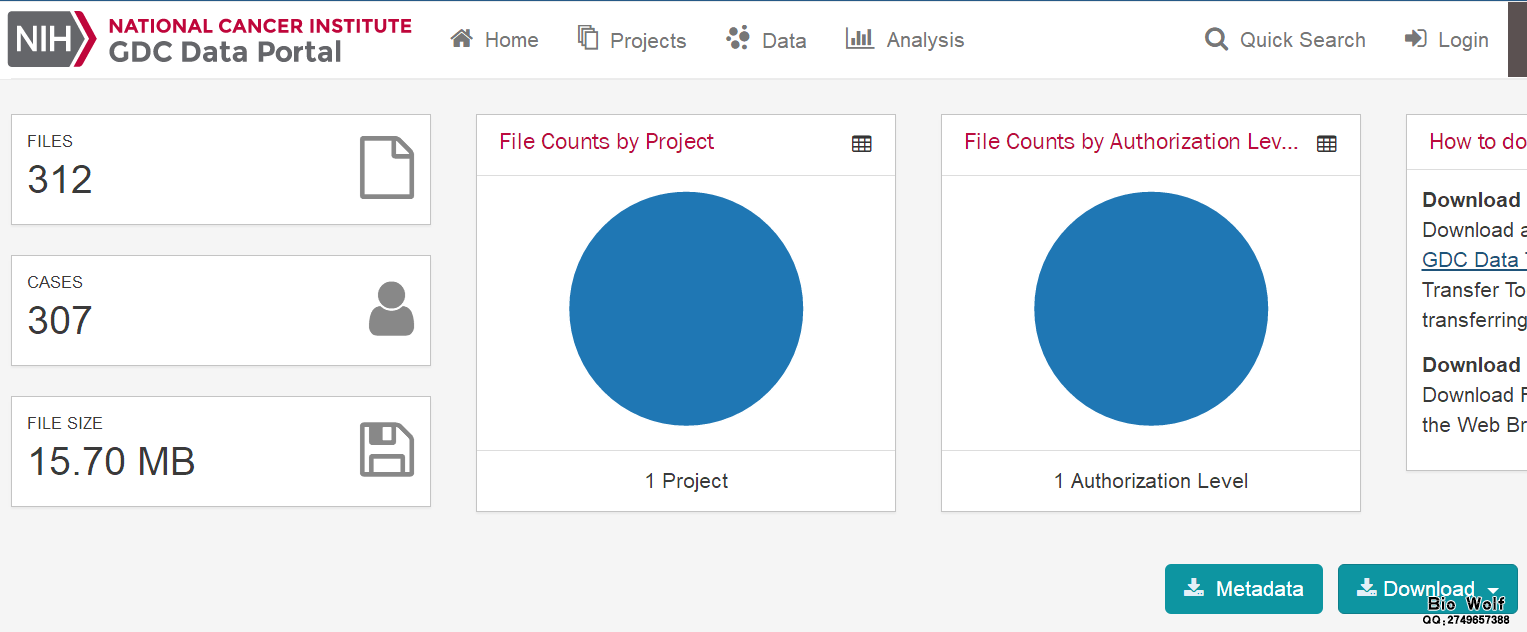 5、当Cart文件夹大于50M时,只能通过Data Transfer Tool工具进行下载。我们这里的Cart是15.70M,可直接可视化下载。  六、提取miRNA矩阵 1、解压Cart压缩包,得到312个文件夹,每个文件夹就是一个数据文件,需要把这些文件提取到一个矩阵文件。 2、把脚本文件,Manifest文件、312个文件夹放在一个文件夹中,运行脚本,即可得到miRNA表达矩阵。 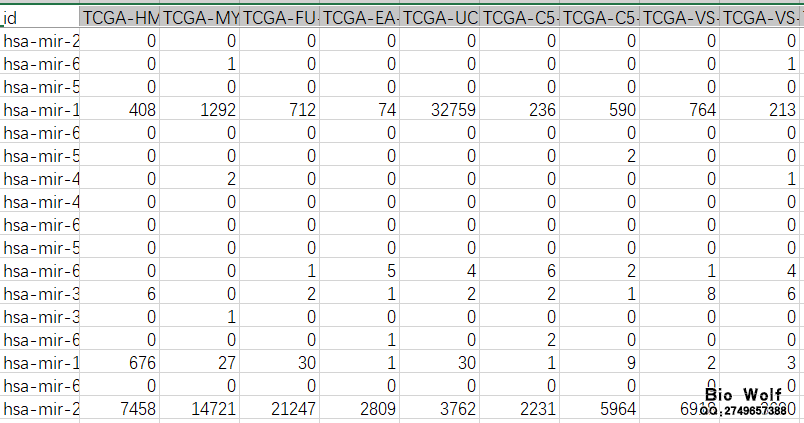

责任编辑:伏泽 作者申明:本文版权属于生信自学网(微信号:18520221056)未经授权,一律禁止转载! |





