|
TCPA数据库筛查预后相关蛋白 1、蛋白表达和生存数据和合并 前面的分析我们得到了两个文件,一个是蛋白表达的文件,一个是临床文件,要做预后筛查分析,就必须通过样本代号把这两份数据合并起来,得到一个新的文件,包含样本代号、蛋白表达、生存时间(单位是年)和生存状态(futime数值1代表死亡,0代表存活) 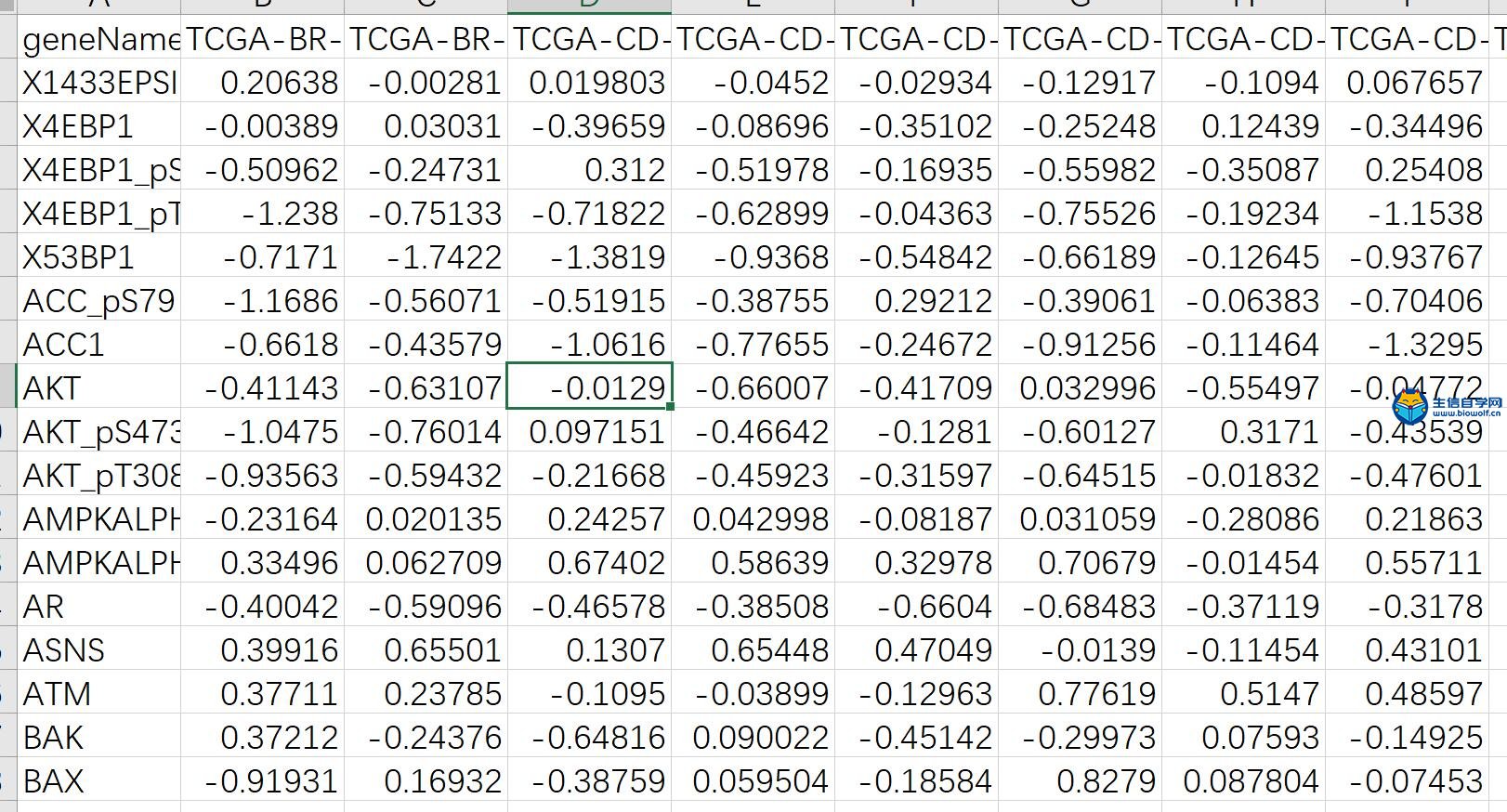 这个输入文件前面已经得到了,只需要用生信自学网专利脚本TCPA07.mergeTime.pl就可以合并,合并之后的数据是这样的: 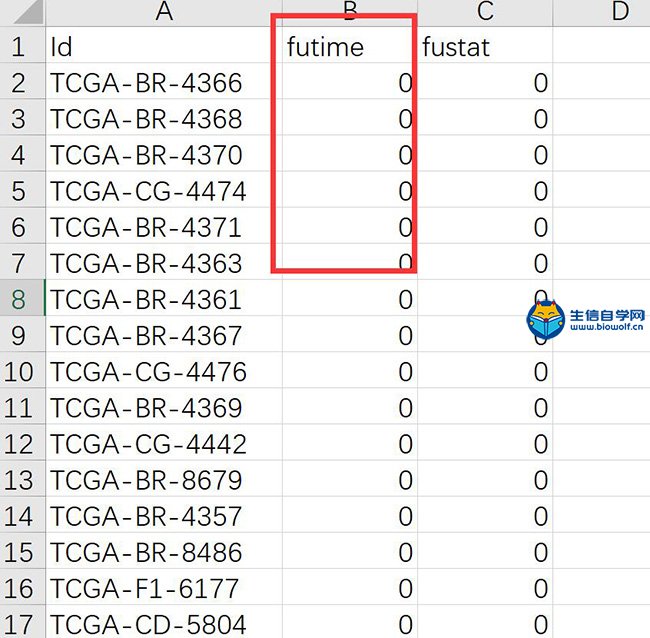 2、预后相关蛋白 蛋白表达数据和生存数据合并,然后用这份数据做预后相关蛋白分析,这里用到了KM方法和Cox方法,那么很多学员希望直到这两种方法的不同 KM方法:根据单个蛋白的表达,把样本分成两组,比较生存是否有差异,可以得到KM方法的P值 Cox方法:把蛋白蛋白表达当成一个连续变量,跟生存时间做比较,看该蛋白表达是否跟生存相关,可以得到HR值,HR值95%CI,Cox方法的P值 这里用到的是R软件做分析,用到的是survival R包,安装方法:install.packages("survival") KM检验,分组标准是蛋白表达量的中位值,函数是:survdiff(),筛选条件p<0.05 单因素cox,用到的函数:coxph,筛选条件p<0.05 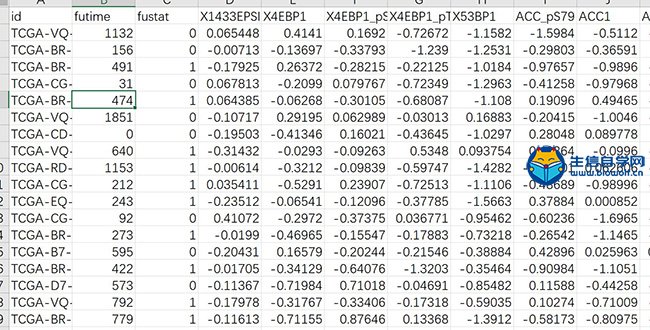 经过处理,可以得到分析的结果文件,以及符合条件蛋白表达文件,用于后续分析 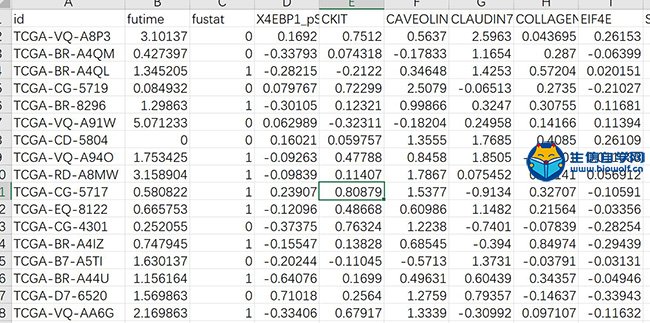 生信自学网精品课程推荐: 《TCGA数据库m6A套路挖掘》 《TCGA单基因文章套路挖掘》 《中药复方网络药理学联合GEO芯片》 
责任编辑:伏泽 作者申明:本文版权属于生信自学网(微信号:18520221056)未经授权,一律禁止转载! |





