|
TCPA数据库预后模型蛋白生存分析生存曲线绘制 输入文件:risk.txt 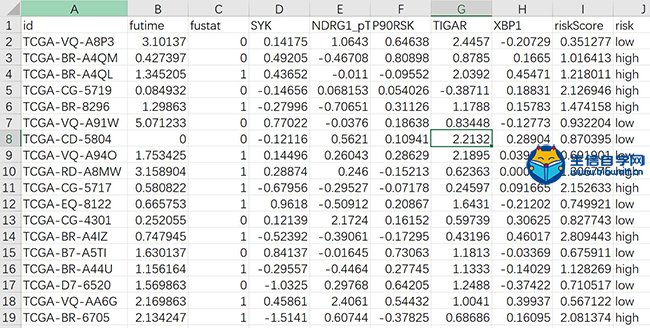 模型蛋白的表达量、生存时间、生存状态 用到的R包: install.packages("survival") install.packages("survminer") 函数:survdiff()、survfit() 分组标准:蛋白表达的中位值,小于等于中位值为低表达组,大于中位值的为高表达组 绘制图形 显示置信区间:conf.int=TRUE 显示图形下半部分风险表格:rusk.table=T 横坐标刻度:break.time.by=1(输入文件futime单位为年,所以这里设定为1) 命令用到for循环,可以一次性绘制所有预后相关蛋白的生存曲线,因为这些蛋白都是经过KM筛选的,所以得到的曲线p值都是小于0.05的。 图形: 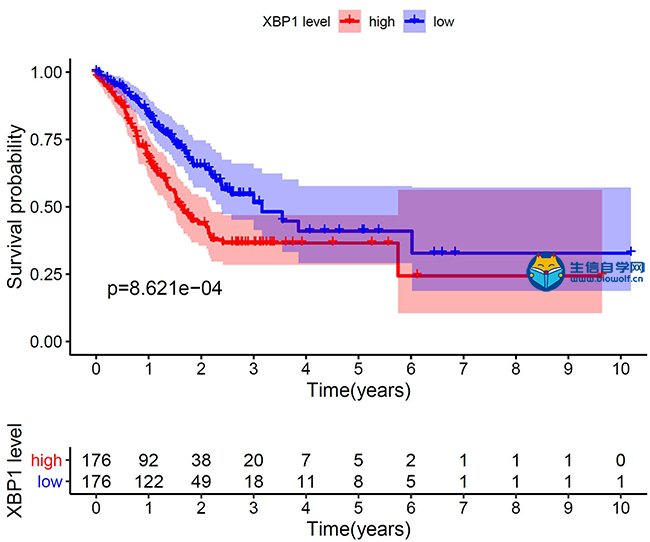 横坐标生存时间(单位是年),纵坐标是生存率,曲线是根据蛋白表达中位值分组,分别是高表达组,低表达组 图形下半部分是生存表格,表格数据是生存时间的样本生存数目,也是分为高低两组 精品课程推荐: 《TCGA蛋白质数据库TCPA挖掘》 《中药复方网络药理学联合GEO》 《单细胞测序分析》 《TCGA单基因发文套路挖掘》 
责任编辑:伏泽 作者申明:本文版权属于生信自学网(微信号:18520221056)未经授权,一律禁止转载! |





